
in Kooperation mit der Universität zu Lübeck
Untersuchungen zum Schilfwachstum an der Wakenitz

Karte
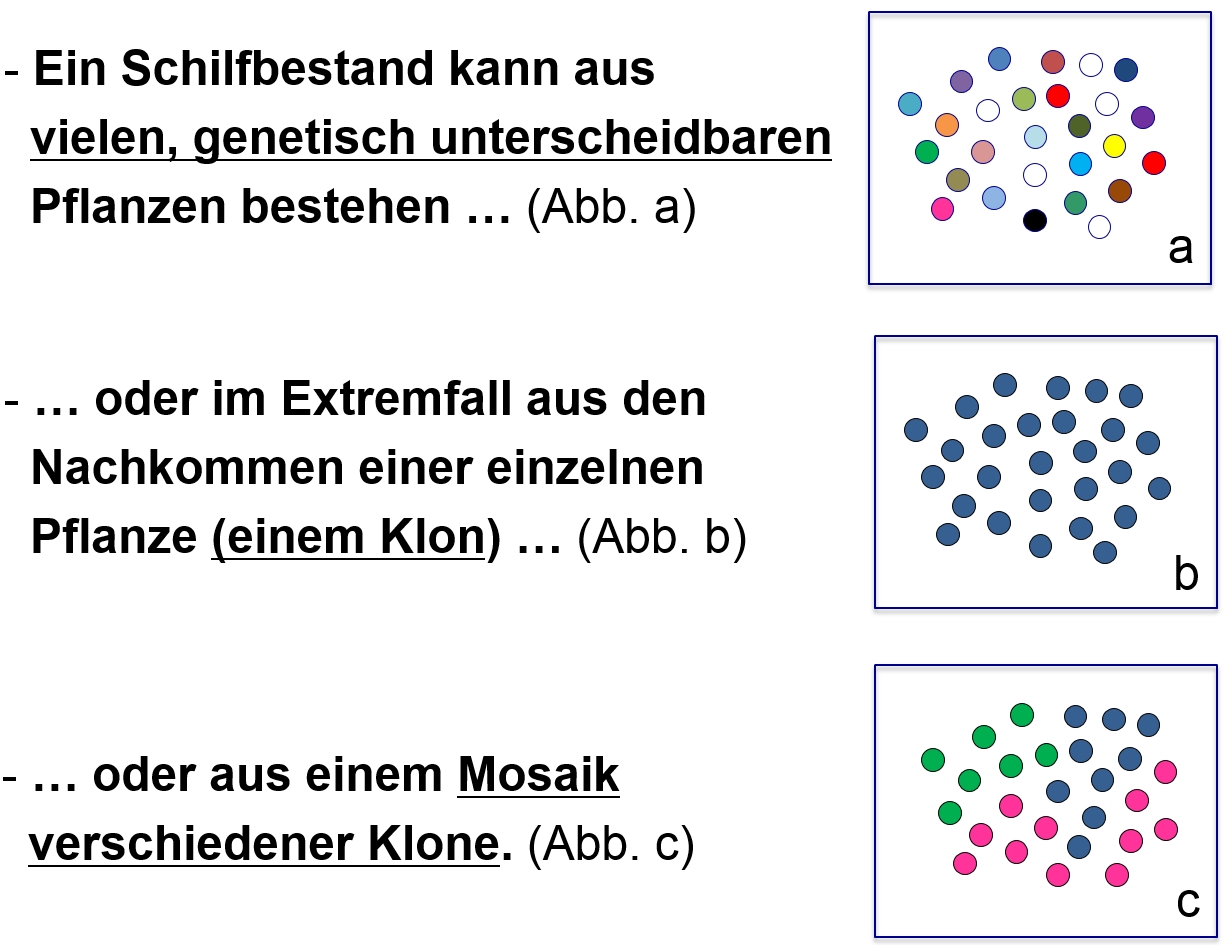
verschiedene Vermehrungswege
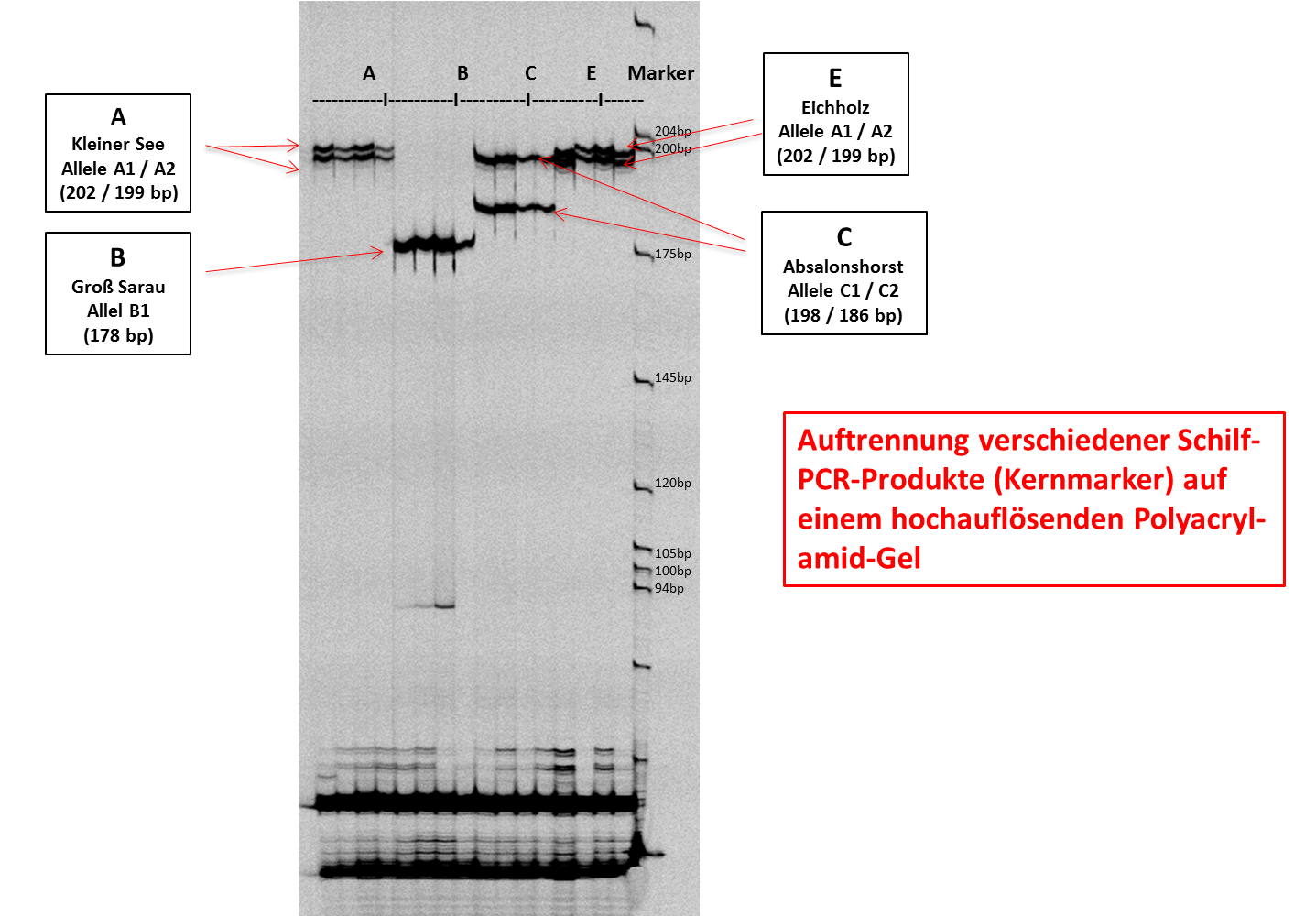
Polyacrylamid-Gel

Agarosegel

Aufarbeitung der Schilfproben; LOLA-Summerschool 2010
Beladen eines Agarosegels; LOLA-Summerschool 2010
Die untersuchten Schilfstandorte an der Wakenitz zeigen im Verlauf der letzten Jahre ein unterschiedliches Wachstumsverhalten. Während die Bestände im Kleinen See und in Eichholz stark rückläufig sind, entwickeln sich die Bestände in Groß Sarau und an der Wallbrechtbrücke (Beprobung seit 2013) gut. Der künstlich angelegte Schilfbestand in Absalonshorst zeigt ein intermediäres Wachstum.
Bisher (Stand: 2013) gibt es keinerlei Hinweise darauf, dass dieses unterschiedliche Wachstumsverhalten eindeutig mit dem Auftreten verschiedener biotischer oder abiotischer Faktoren zusammen hängt.
Es ist also denkbar, dass dieses unterschiedliche Wachstumsverhalten das Auftreten unterschiedlicher Genotypen wiederspiegelt. D.h. auf ähnliche Umweltbedingungen reagieren die Bestände, aufgrund ihrer unterschiedlichen genetischen Konstitution, verschieden. Diese Vermutung wird durch die besondere Vermehrungsweise des Schilfrohrs untermauert.
Schilf kann sich sowohl generativ (über Samen) als auch vegetativ (über Rhizomsprosse) vermehren. Das kann zu verschiedenen genetischen Szenarien führen (s. Abb. rechts):
Klonales Wachstum kann sehr vorteilhaft sein: Unter günstigen Umweltbedingungen können schnell große Flächen besiedelt werden, ohne die Ausbildung von Blüten und Samen. Auf der anderen Seite kann klonales Wachstum bei geänderten Umweltbedingungen fatale Folgen haben: Ist der vorliegende Genotyp nur unzureichend angepasst, ist davon der gesamte Bestand betroffen.
Zur Testung der Schilfgenotypen wurde an den verschiedenen Standorten junges Pflanzenmaterial gesammelt und jeweils ein genetischer Fingerabdruck angefertigt. Diese Technik entspricht dem genetischen Fingerabdruck, wie er in der Forensik angewendet wird. Mit dieser Technik werden genetische Unterschiede zwischen verschiedenen Individuen einer Art analysiert. Aussagekräftige Unterschiede findet man innerartlich nur in nicht-kodierenden Bereichen der DNA. Das können sowohl Introns als auch intergenische Sequenzabschnitte sein. Da Mutationen in diesen DNA-Bereichen keinerlei Konsequenzen für den Phänotyp haben, gibt es keinen Selektionsdruck gegen diese Sequenzveränderungen, sodass sie sich in diesen nicht-kodierenden Bereichen anhäufen.
In diesem Schilfprojekt werden solche Unterschiede mittels Polymerase-Kettenreaktion (PCR) und anschließender Gel-Elektrophorese analysiert. Diese Technik ist nur dann aussagekräftig, wenn die PCR-Produkte unterschiedliche Größen aufweisen; die Mutationen also zu einem Verlust oder einem Zugewinn an DNA-Sequenz geführt haben. Solche Veränderungen treten gehäuft im Bereich von Mikrosatelliten auf, also Clustern aus kurzen, vielfach wiederholten DNA-Sequenzen. Flankieren die beiden PCR-Primer ein solches Cluster, kann dessen variable Grösse bestimmt werden (s.u.).
| Forward Primer | ....... (TAC)n ....... | |
| ....... (ATG)n ....... | Backward Primer |
Verändert sich der Repetitionsgrad "n", führt dies zu unterschiedlichen Größen der PCR-Produkte.
Das gesammelte junge Pflanzenmaterial wurde bis zur Bearbeitung bei -80 °C gelagert. Zur DNA-Isolation wurde das Pflanzen-DNA-Isolations-Kit der Firma Qiagen verwendet (www.qiagen.com). Dieser Kit kam erstmals in der LOLA-Summerschool 2010 zum Einsatz. Im Vorfeld wurde die Herstelleranleitung von Herrn PD Dr. C.L. Schmidt auf das Silikat-haltige Schilfmaterial adaptiert, sodass die Versuchsanleitung final 33 Arbeitsschritte umfaßte, statt der ursprünglichen 12 Arbeitsschritte.
Qualität und Quantität der isolierten DNA-Proben wurden auf einem 1%-igen, Ethidiumbromid-gefärbten Agarosegel überprüft.
Im Folgenden sind die aussagekräftigen Ergebnisse von drei Primer-Paaren dargestellt; ein Kernmarker, zwei Chloroplastenmarker (DNA-Sequenzen:https://www.ncbi.nlm.nih.gov/genbank/).
Die PCR-Produkte wurden entweder mit hochauflösenden Polyacrylamid-Gelen analysiert (Trennschärfe ± 3 Basenpaare, (s. Abb. rechts)) oder mit hochprozentigen Agarosegelen (Trennschärfe ± 10 Basenpaare, (s. Abb. rechts)). Ein Teil der PCR-Produkte wurde kommerziell sequenziert.
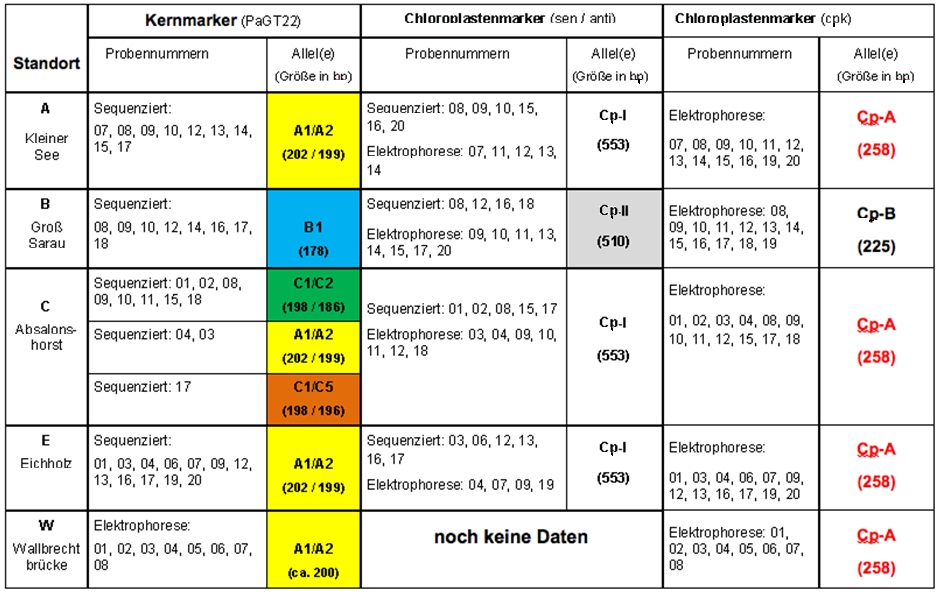
Tabellarische Zusammenstellung der genetischen Schilf-Daten (Stand: Dezember 2013)
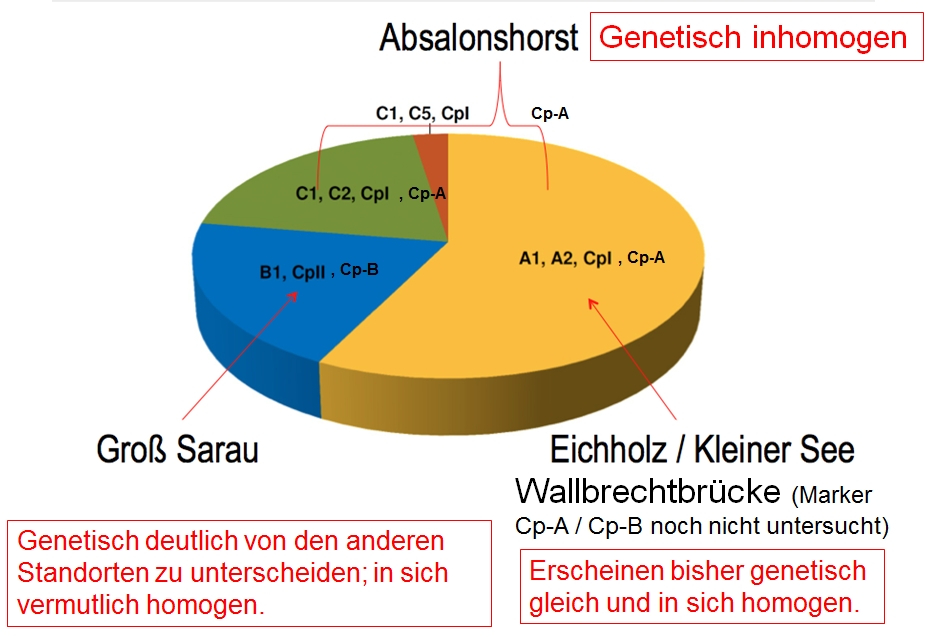
Grafische Zusammenstellung der genetischen Schilf-Daten (Stand: Dezember 2013)
Die Schilfbestände an den Standorten Eichholz und Kleiner See erscheinen bisher genetisch gleich und in sich homogen. Kern-lokalisiert wurden die Allele A1 und A2 nachgewiesen, Chloroplasten-lokalisiert die Allele Cp-I und Cp-A. Auch am Standort Wallbrechtbrücke, der seit 2013 beprobt wird, wurde bisher ausschließlich dieser Genotyp gefunden; allerdings wurden dort bisher erst zwei Marker getestet (es fehlt noch Cp-A / Cp-B).
Dieser Genotyp (A1 / A2, Cp-I, Cp-A) wurde auch am Standort Absalonshorst gefunden, jedoch zusätzlich zwei weitere Kern-lokalisierte Allel-Kombinationen ( C1 / C2 und C1 / C5). Diese Genotypen tragen im Chloroplasten wiederum die Allele Cp-I und Cp-A. Die Heterogenität der Genotypen in Absalonshorst ist möglicherweise die Folge der künstlichen Schilf-Anpflanzung an diesem Standort.
Genetisch gut zu unterscheiden von den vier oben beschriebenen Beständen sind die analysierten Schilfproben vom Standort Groß Sarau. Die 12 untersuchten Proben sind untereinander homogen und klar zu unterscheiden von den beschriebenen Genotypen an den anderen Standorten. Der Genotyp "Groß Sarau" ist in den Abbildungen blau markiert und beinhaltet das Kern-lokalisierte Allel B1 (homozygot) und die Chloroplasten-lokalisierten Allele Cp-II und Cp-B.
Nach den derzeitigen Untersuchungen dominiert der Genotyp "A1 / A2, Cp-I, Cp-A". Er wurde in allen Proben vom Kleinen See und von Eichholz gefunden, sowie in wenigen Proben aus Absalonshorst. Sollte dieser Genotyp anfällig sein für veränderte Umweltbedingungen, könnte dies den starken Rückgang der Schilfbestände am Kleinen See und in Eichholz erklären, sowie das intermediäre Wachstum am Misch-Standort Absalonshorst.
In allen acht Proben des gut wüchsigen Standorts Wallbrechtbrücke wurden bisher ebenfalls die Allele A1 / A2 und das Allel Cp-I nachgewiesen; der Chloroplasten-marker Cp-A / Cp-B wurde bisher noch nicht getestet. Sollte sich auch hier ausschliesslich das Allel Cp-A finden, kann der Genotyp "A1 / A2, Cp-I, Cp-A" nicht allein verantwortlich sein für den Schilfrückgang an den Standorten Kleiner See und Eichholz. Da die vorliegende Analyse, mit derzeit drei untersuchten Markern, jedoch sehr punktuell ist, kann nicht ausgeschlossen werden, dass sich der Genotyp am Standort Wallbrechtbrücke von den Genotypen Kleiner See und Eichholz unterscheidet, die Unterschiede bisher lediglich nicht erfaßt wurden.
Klar ist hingegen der genetische Unterschied zum Standort Groß Sarau. Dort hatten alle 11 untersuchten Proben den abweichenden Genotyp "B1, Cp-II, Cp-B". Dieser Genotyp wurde bisher an keinem der anderen Standorte gefunden. Somit könnte dieser Genotyp mit einem widerstandsfähigen Phänotyp einhergehen, der das gute Schilfwachstum in Groß-Sarau ermöglicht.
Vervollständigung der genetischen Analyse der Proben vom Standort Wallbrechtbrücke.
Ob der in Groß Sarau nachgewiesene Genotyp ursächlich für das gute Schilfwachstum an diesem Standort verantwortlich ist, müssten Verpflanzungsexperimente dieser Rhizome an einen anderen Standort zeigen. In Groß Sarau entnommene Rhizome befinden sich derzeit unter kontrollierten Zuchtbedingungen und zeigen den Sarau-typischen Genotyp. Dieser spezielle Genotyp ist mit Hilfe der oben beschriebenen Techniken auch nach einer Verpflanzung jederzeit wieder auffindbar, sofern sich die Pflanzen rein vegetativ vermehren.